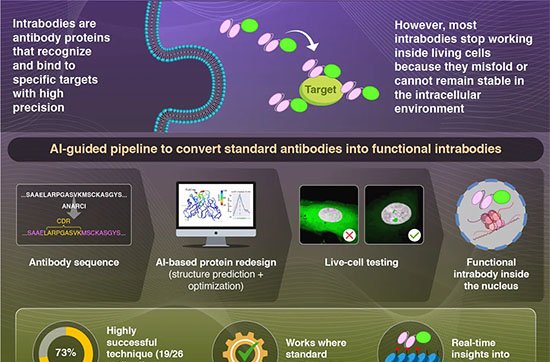
Pesquisadores da Universidade de Tecnologia de Graz (TU Graz), na Áustria, apresentaram um método que combina inteligência artificial e dados evolutivos para localizar, com alta precisão, quais aminoácidos controlam a função e a estabilidade de proteínas. O trabalho, liderado por Andreas Winkler e Oliver Eder, foi publicado em 30 de outubro de 2025 na revista Structure.
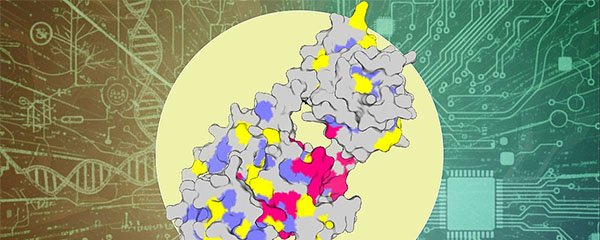
Denominada Function-Structure-Adaptability (FSA), a abordagem compara sequências geradas pelo modelo de deep learning ProteinMPNN — projetado para criar cadeias de aminoácidos capazes de adotar uma estrutura tridimensional estável — com sequências naturais preservadas ao longo de milhões de anos. A análise indica se um aminoácido é essencial para a atividade biológica da proteína ou para manter sua conformação.
Como sistema-teste, a equipe utilizou a família de bacteriofitocromos, fotossensores bacterianos envolvidos na detecção de luz. Quando um resíduo aparece repetidamente em proteínas naturais, mas não é destacado pelo ProteinMPNN, ele tende a ter papel funcional; se surge nos dois conjuntos, é considerado estrutural.
Aminoácidos classificados em três grupos
Para viabilizar a comparação estatística, os cientistas agruparam os 20 aminoácidos conforme suas propriedades químicas, permitindo dividi-los em três categorias:
- Funcionais – determinam a atividade específica da proteína;
- Estruturais – garantem estabilidade e dobramento corretos;
- Adaptáveis – grupo intermediário que requer estudos adicionais.
Ensaios de laboratório confirmaram as previsões. Alterações dirigidas nos resíduos classificados impactaram significativamente a resposta à luz dos bacteriofitocromos, e os acertos coincidiram com dados já descritos na literatura.

Imagem: Internet
Segundo Oliver Eder, análises que antes podiam levar meses agora podem ser iniciadas em cerca de uma semana, graças ao pré-filtro fornecido pela FSA. A metodologia é aplicável a qualquer classe de proteína, abrindo caminho para o desenvolvimento de fármacos, otimização de enzimas industriais e compreensão de mecanismos como a resistência a antibióticos.
Com informações de Nanowerk