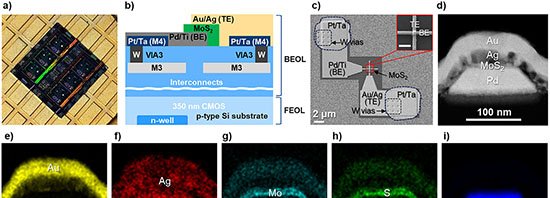
Engenheiros da Universidade da Pensilvânia apresentaram um arcabouço matemático capaz de converter movimentos atômicos e moleculares em previsões sobre comportamentos macroscópicos, como o desdobramento de proteínas, a formação de cristais e o derretimento do gelo, sem recorrer a simulações ou experimentos demorados e onerosos.
O método, batizado de Stochastic Thermodynamics with Internal Variables (STIV), foi descrito em artigo recente do Journal of Non-Equilibrium Thermodynamics. A abordagem permitiu solucionar um impasse de 40 anos na modelagem de campo de fases, ferramenta usada para estudar a fronteira entre dois estados da matéria, como a interface água–gelo ou a junção entre regiões dobradas e desdobradas de uma proteína.
“A modelagem de campo de fases busca prever o que ocorre nesse limite fino entre fases. O STIV nos dá a estrutura matemática para descrever essa evolução a partir de primeiros princípios, sem ajustes empíricos”, afirmou o professor Prashant Purohit, do Departamento de Engenharia Mecânica e Mecânica Aplicada (MEAM), coautor do estudo.
Três métodos para diferentes cenários
No trabalho mais recente, os pesquisadores ampliaram o alcance do STIV, apresentando três métodos capazes de abranger praticamente qualquer situação fora do equilíbrio. Dois deles entregam resultados mais rápidos e cobrem a maioria dos sistemas; o terceiro exige maior tempo de cálculo, mas lida com casos raros, explicou o primeiro autor Travis Leadbetter, doutor recém-formado em Matemática Aplicada e Ciência Computacional.
Do átomo ao material
Segundo a professora Celia Reina, autora sênior da pesquisa, o STIV funciona como uma “Pedra de Roseta” porque estabelece uma correspondência entre variáveis internas escolhidas e o comportamento em escala maior. A escolha adequada dessas variáveis é essencial para que a matemática produza previsões confiáveis sem ajustes adicionais.

Imagem: Internet
A equipe destaca que a nova estrutura poderá acelerar o desenvolvimento de medicamentos, semicondutores e outros materiais, já que permite partir da propriedade desejada e retroceder até os movimentos moleculares responsáveis por ela. Pesquisadores nos Estados Unidos e na Itália já aplicaram o STIV para investigar a locomoção de células biológicas, demonstrando a versatilidade da ferramenta.
Com informações de Nanowerk