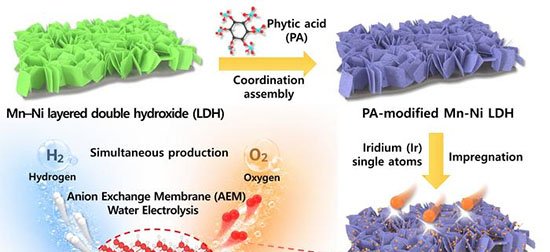
Uma nova plataforma baseada em nanoporos sólidos conseguiu transformar sinais bioquímicos em respostas digitais, abrindo caminho para computação de DNA escalável e biossensores sem uso de corantes. O avanço foi descrito em artigo publicado em 28 de outubro de 2025 na revista Advanced Functional Materials.
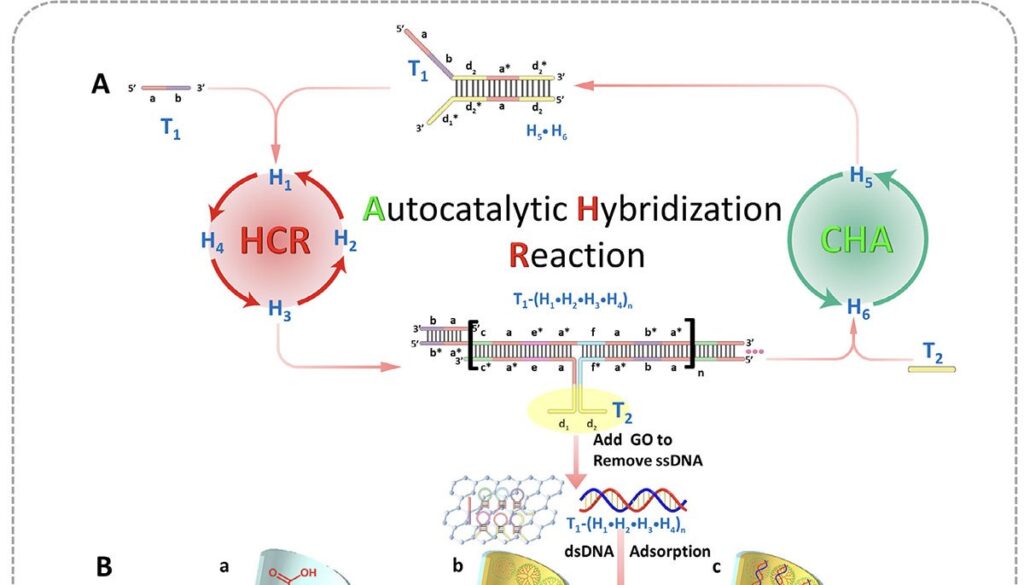
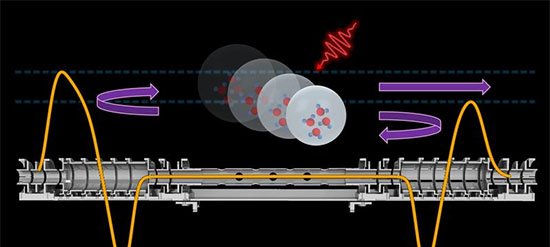
O sistema combina nanoporos cônicos de polímero, revestidos internamente com o dendrímero carregado positivamente G4-PAMAM, a um circuito de DNA que amplifica sinais em duas direções. Essa arquitetura permite detectar pequenas variações de corrente elétrica e executar operações lógicas padrão sem qualquer modificação adicional no dispositivo.
Como funciona o circuito
No centro da tecnologia está a reação de hibridação autocatalítica (AHR), que integra duas rotas conhecidas de amplificação isoterma — reação em cadeia por hibridação (HCR) e montagem catalítica de grampos (CHA). Ambas operam à temperatura ambiente e dispensam enzimas.
Dois gatilhos, T1 e T2, podem iniciar a AHR. T1 abre uma sequência de grampos H1–H4, formando longos “fios” de DNA. Ao mesmo tempo, esses eventos recompõem T2, que ativa outro conjunto de grampos (H5 e H6) e produz múltiplos fragmentos curtos. Os produtos liberam novos T1, fechando um ciclo de realimentação que gera grandes quantidades de DNA carregado negativamente.
Ao aderirem à superfície revestida de PAMAM, esses fios neutralizam a carga positiva do poro e reduzem a retificação de corrente iônica. O resultado é uma queda estável de corrente, medida em tensão fixa, menos suscetível a ruídos do que os picos transitórios normalmente observados em translocações individuais.
Detecção de polinucleotídeo quinase
Para demonstrar aplicação prática, os pesquisadores criaram um ensaio para polinucleotídeo quinase (PNK), enzima crucial na reparação de DNA. Um grampo projetado (HPNK) esconde o gatilho T1. Primeiro, a PNK fosforila o grampo; em seguida, a exonuclease lambda digere a extremidade fosforilada, liberando T1 e acionando a cascata.
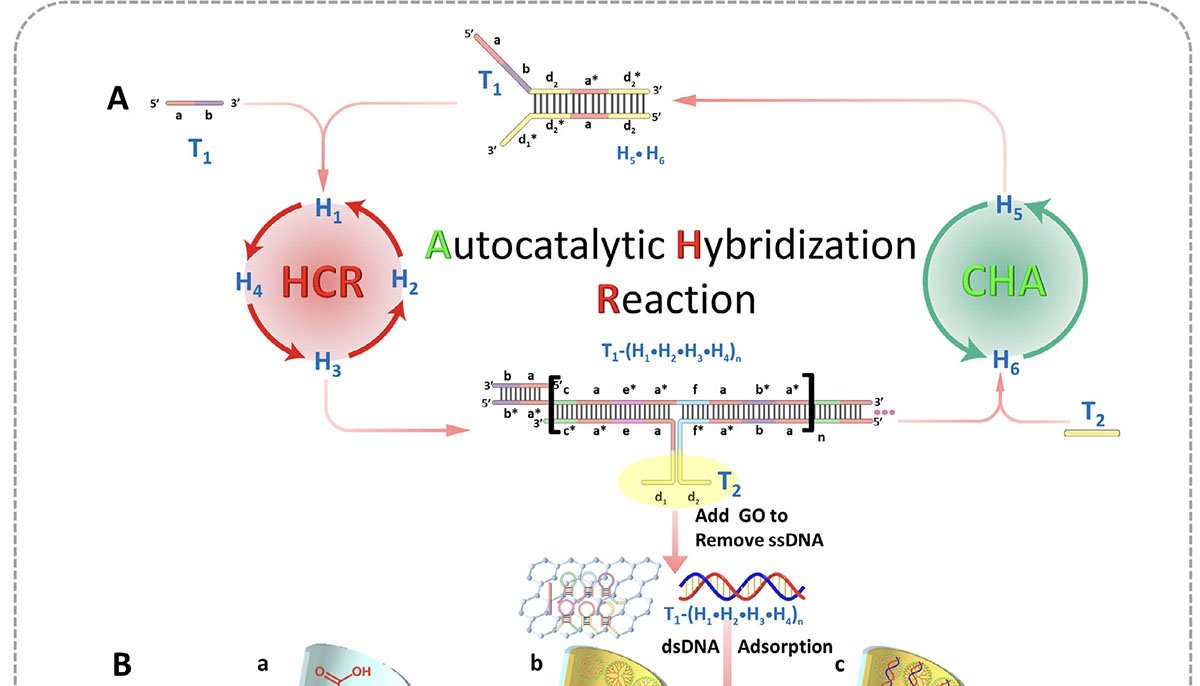
Imagem: coupling autocatalytic amplificati wit
O sinal apresentou correlação linear (R² = 0,9982) entre a intensidade de corrente e o logaritmo da concentração de PNK, em faixa de 0,0001 a 0,2 U/mL. O limite de detecção foi de 0,0001 U/mL. Todo o procedimento — incubação enzimática, amplificação e remoção de fitas não reagidas com óxido de grafeno — dura cerca de 190 minutos.
Lógica molecular em um único poro
A mesma configuração executou seis portas Booleanas: OR, YES, AND, INHIBIT, NOR e NAND. A presença ou ausência de fitas de DNA específicas funciona como entrada, enquanto a corrente, comparada a um limiar fixo de 40 nA, define a saída (1 ou 0). Todas as operações usam os mesmos componentes de DNA, dispensando reconfiguração do nanopo.
Segundo os autores, o método mantém vantagens práticas: amplificação livre de enzimas, temperatura constante, tampões padrão e leitura elétrica direta, fatores que favorecem dispositivos portáteis de baixo consumo.
Com informações de Nanowerk